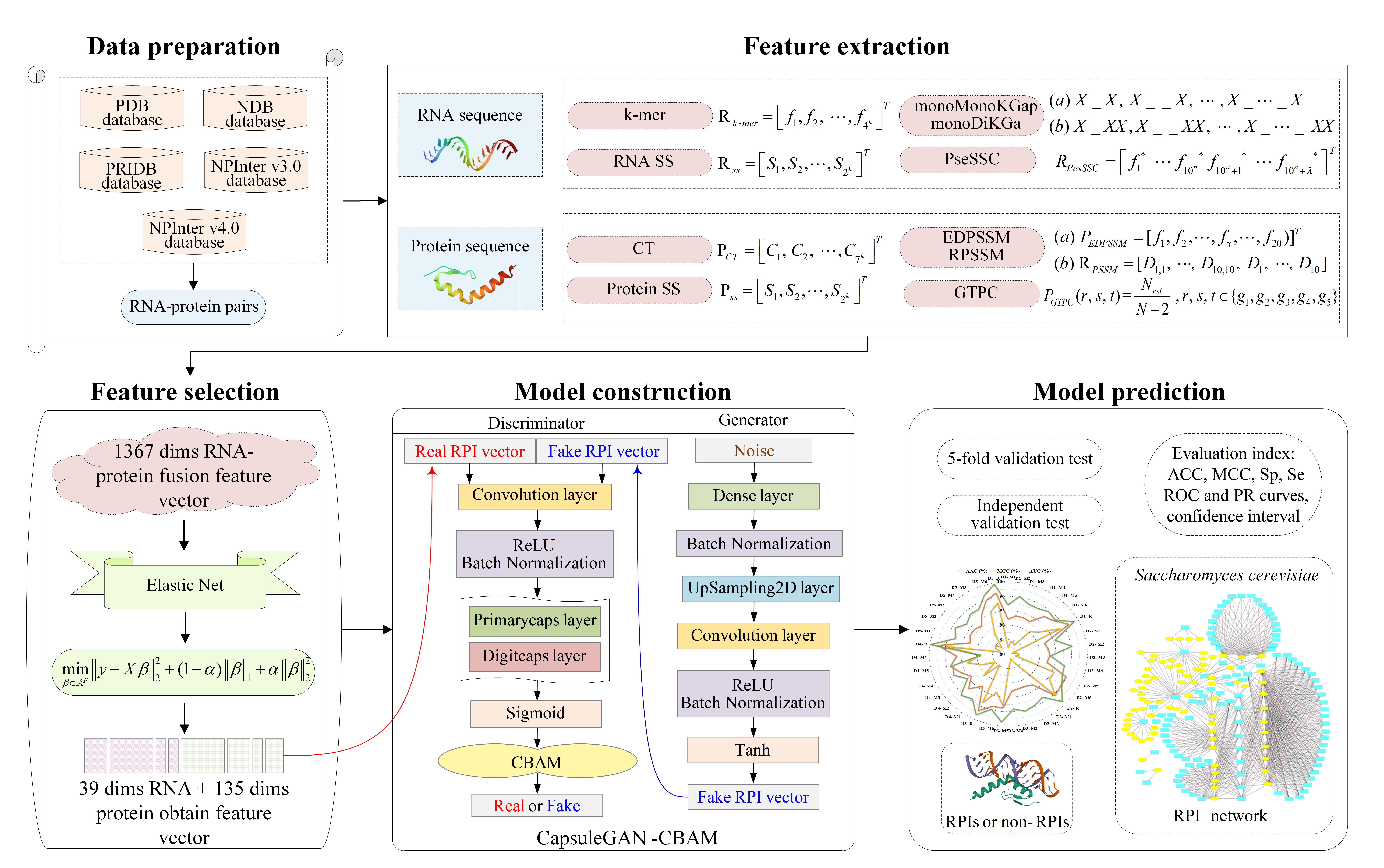
近日,学校信息学院人工智能与生物医学大数据研究团队于彬教授在人工智能领域的顶级期刊Pattern Recognition (IF=8.518)上发表标题为“RPI-CapsuleGAN: Predicting RNA-protein interactions through an interpretable generative adversarial capsule network”的研究论文,构建预测RNA-蛋白质相互作用的人工智能算法模型—RPI-CapsuleGAN。该模型表现出较强的泛化能力以及模型预测的稳定性和鲁棒性。于彬教授为论文的唯一通讯作者,研究生王逸飞、王雪并列第一作者,262net必赢为第一完成单位。
本研究首次将卷积注意力机制引入生成对抗胶囊网络构建RNA-蛋白质相互作用预测模型框架。其中,生成对抗胶囊网络中生成器和判别器两个模型经过不断博弈,可有效解决训练过程中存在的样本数据量不足、稳定性不够以及模型空间结构层次消失等问题。卷积注意力机制通过为每个输入特征分配权重优化特征空间,可配合生成对抗胶囊网络算法进一步提高模型的可解释性,实现对RPI更高效、更精准、更稳定的预测。在5折交叉验证测试中,RPI-CapsuleGAN模型在数据集RP1488、RPI369、RPI2241、RP11807和RPI1446上优于其它现有RPI预测算法。六个不同物种的独立测试集结果表明基于生成对抗胶囊网络的深度学习算法具有较好的模型泛化能力。本文提出的RPI-CapsuleGAN模型,不仅为探索数以百万计的潜在RNA-蛋白质相互作用对提供一定见解和参考,同时也为相关性疾病机理研究提供重要线索和理论依据,有望为新药研发和疾病治疗提供指导。
文章链接:https://doi.org/10.1016/j.patcog.2023.109626
此外,该团队近一年来基于人工智能算法在生物信息学、人工智能领域的高水平SCI期刊Bioinformatics、Knowledge-Based Systems、Applied Soft Computing取得四项关于蛋白质组学研究的重要科研成果,相关工作已发表论文:
Predicting the multi-label protein subcellular localization through multi-information fusion and MLSI dimensionality reduction based on MLFE classifier. Bioinformatics, 2022, 38(5): 1223-1230 (IF=6.931 CCF B)。于彬教授为论文的唯一通讯作者,刘玉霜副教授第一作者。
Malsite-Deep: Prediction of protein malonylation sites through deep learning and multi-information fusion based on NearMiss-2 strategy. Knowledge-Based Systems, 2022, 240: 108191 (IF=8.139 TOP)。于彬教授为论文的唯一通讯作者,王明辉教授第一作者。
DEEPStack-RBP: Accurate identification of RNA-binding proteins based on autoencoder feature selection and deep stacking ensemble classifier. Knowledge-Based Systems, 2022, 256: 109875 (IF=8.139 TOP)。于彬教授为论文的唯一通讯作者,研究生韦芹芹、张青梅并列第一作者。
RPI-MDLStack: Predicting RNA-protein interactions through deep learning with stacking strategy and LASSO. Applied Soft Computing, 2022, 120: 108676 (IF=8.263 TOP)。于彬教授为论文的第一作者及通讯作者,研究生王雪第二作者。
以上四篇论文均以262net必赢为第一完成单位。此项研究得到了国家自然科学基金面上项目(62172248)和山东省自然科学基金面上项目(ZR2021MF098)的资助。






